Neues Gen für Coffin-Siris-Syndrom gefunden
Das Coffin-Siris-Syndrom (CSS) ist eine schwere Form der psychomotorischen Entwicklungsstörung. Ursache sind Neumutationen in einzelnen Mitgliedern des BAF-Chromatin-Remodeling-Komplexes. Die Arbeitsgruppe von Prof. Reis hat nun eine weitere genetische Ursache für dieses Syndrom identifiziert.
Im BMBF geförderten Forschungs-Netzwerk Chromatin-Net beschäftigt sich die Gruppe mit CSS und anderen seltenen Erkrankungen, die mit einer gestörten Chromatin-Dynamik assoziiert sind. In einem Artikel im renommierten American Journal of Human Genetics berichtet die Gruppe nun über Neumutationen im DPF2-Gen (double plant homeodomain finger 2), welches für eine BAF-Untereinheit kodiert und bisher mit keiner Erkrankung in Zusammenhang gebracht wurde.
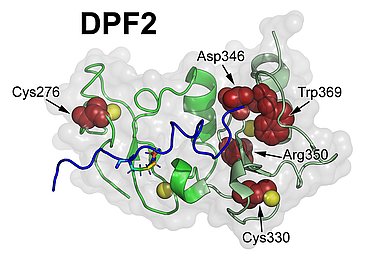
Bei insgesamt 8 Patienten mit typischen klinischen Merkmalen des CSS identifizierten sie jeweils heterozygote DPF2-Neumutationen. Die Hauptsymptome sind eine globale Entwicklungsverzögerung, variable kognitive Einschränkungen, eine ausgeprägte Sprachstörung und verschiedene körperliche Auffälligkeiten. Charakteristisch ist außerdem das Fehlen oder eine Unterentwicklung der Fingernägel und/oder Zehennägel. In umfangreichen funktionellen in vitro Studien konnte die Gruppe eine durch die Mutationen bedingte Beeinträchtigung der Strukturintegrität und Kohäsion der PHD-Fingerdomänen von DPF2 zeigen, die wiederum zu einer gestörten Erkennung von Histonmodifikationen führt und somit zu einer beeinträchtigten Genregulation. Auf Grund der Daten vermuten die Forscher außerdem einen dominant-negativen Mutationsmechanismus. Diese Studie unterstreicht die Rolle gestörter Chromatin Regulation in der Entstehung verschiedener psychomotorischer Entwicklungsstörungen sowie den Zusammenhang von Mutationen des BAF-Komplexes mit der Ätiologie des CSS.
Der BAF-Komplex, auch bekannt als SWI/SNF-A-Komplex, ist ein Multiproteinkomplex, der an der Aktivierung oder Repression der Gentranskription durch die Modifikation der Nukleosomenstruktur beteiligt ist. Nukleosomen sind die Bausteine des Chromatins, der funktionellen Form der DNA im Zellkern. Dabei ist die DNA um ein Histonoktamer aus je zwei Kernhistonen H2A, H2B, H3 und H4 gewunden. DPF2 enthält zwei PHD (plant homeodomain) Domänen, die spezielle Histonmodifikationen, wie Methylierung und Acetylierung erkennen. Proteine mit PHD-Fingern spielen eine wichtige Rolle in der Transkriptionsregulation. Alle identifizierten DPF2-Mutationen waren innerhalb der evolutionär hoch konservierten PHD1- und PHD2-Zinkfingerdomäne lokalisiert. In Zusammenarbeit mit der Arbeitsgruppe von Prof. Felix Engel (Nephropathologische Abteilung) führte die Arbeitsgruppe umfangreiche funktionelle Studien durch. So konnten sie für einzelne Mutationen eine beeinträchtigte Bindung an unmodifizierte und modifizierte H3-Histone nachweisen, während die Interaktion mit H4-Histonen unverändert blieb. Expression mutierter DPF2-Proteine führte zur Entstehung von Aggregaten des BAF-Komplexes im Zellkern verschiedener Zelllinien, entsprechend dem dominant-negativen Mutationsmechanismus. Die durch Mutationen veränderte Proteinkonformation und gestörte Chromatin-Erkennung führt zu einer beeinträchtigten Genregulation. Wie genau diese Störungen der Zellfunktion dann zu Entwicklungsstörungen führen, muss in weiteren Studien untersucht werden.
Kontakt:
Prof. Dr. André Reis
E-Mail: andre.reis(at)uk-erlangen.de