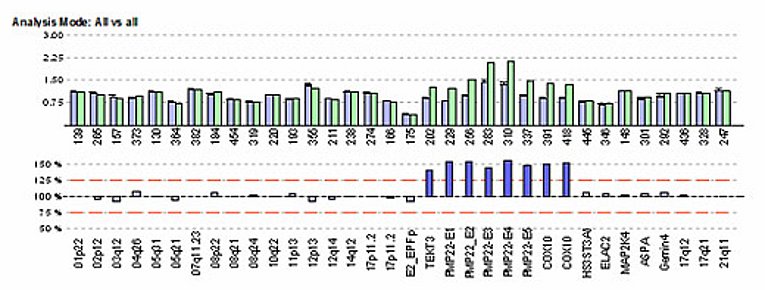
Nachweis von größeren Deletionen/Duplikationen mittels MLPA
Die MLPA Methode („Multiplex Ligation-dependent Probe Amplification“) ermöglicht die Bestimmung von Dosisunterschieden von Genen und einzelnen Genabschnitten (Exons).
Diese Methode zur Detektion von Kopienzahlveränderungen (Deletionen/ Duplikationen) in nur einem Ansatz mit bis zu 50 verschiedenen DNA-Fragmenten wurde erstmals 2002 beschrieben (Schouten et al., Nucl Acids Res 30:12e57). Die MLPA-Reaktion erfolgt im Wesentlichen in vier Schritten: (1) DNA-Denaturierung und Hybridisierung der MLPA-Proben, (2) Ligationsreaktion, (3) PCR und (4) Trennung der Amplifikationsprodukte durch Kapillarelektrophorese (ABI3500xl, ABI3730, Thermo Fisher Scientific).
Kopienzahlveränderungen sind durch Vergrößerung bzw. Reduktion der Peakhöhen/-flächen im Vergleich zu den Kontrollen erkennbar. Jede der bis zu 50 MLPA-Proben in einem Ansatz besteht aus einer “Target”-spezifischen Sequenz (Hybridisierungssequenz), einer “Stuffer” (Fragmentlängen)-Sequenz definierter Länge und einer M13-Universal-Primersequenz. Die Multiplex-Amplifikation mit einem Fluoreszenz-markierten M13-Primerpaar kann nur nach Ligation der hybridisierten Proben erfolgen. Durch das Design der unterschiedlich langen “Stuffer”-Fragmente ist eine Trennung von Fragmenten einer Länge zwischen 130 und 480 bp möglich.
Für die MLPA werden Kits von MRC Holland verwendet (https://www.mrcholland.com). Die Auswertung erfolgt mit dem MLPA® Modul der Sequence Pilot Software (JSI medical Systems; jsi-mediysy.de):